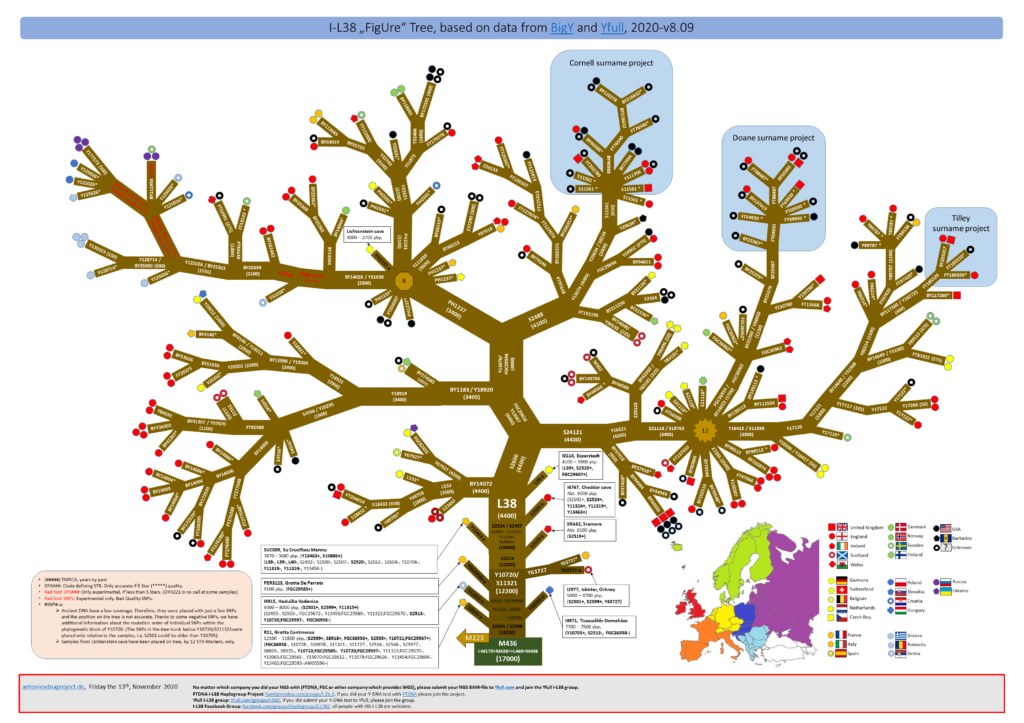
Diesen I-L38 – FigUre Baum gibt es auch als PDF zum Runterladen unter: I-L38 – FigUre tree.pdf, oder als I-L38 – FigUre tree.png mit hoher Auflösung.
Diese Darstellung des I-L38 Baumes vereint die Haplobäume von FTDNA’s Big Y und dem Baum von YFull.com. Es wurden fast nur NGS (Next Generation Sequencing) aufgenommen. Tester, welche nur Einzel SNPs getestet haben, wurden nur dann aufgenommen ((SNP-P) #SNP#), wenn alle SNPs im unmittelbaren Umfeld getestet wurden, so dass die genaue Platzierung gewehrleistet ist.
Zusätzlich wurden alle momentan bekannten aDNA (ancient DNA) Proben mit aufgenommen (#SNP#-a). Diese haben eine geringe Abdeckung. Daher wurden sie anhand weniger SNPs platziert und die Position auf dem Baum ist nicht genau. Dank einiger negativer SNPs haben wir zusätzliche Informationen über die Mutationsreihenfolge einzelner SNPs, innerhalb des phylogenetischen Blockes von Y10705. (Die SNPs im Baumstamm unterhalb Y10705/S2505 wurden nur bezogen auf die Proben platziert, d.h. Y10705 könnte älter sein als S2501). Die Proben aus der Höhle Lichtenstein wurden nur anhand von 12 STR-Markern auf den Baum platziert.
Die Zahlen in Klammern unterhalb der SNPs in den einzelnen Ästen geben den TMRCA (Time to Most Recent Common Ancestor) an. Diese wurden der Age estimation von YFull.com entnommen. Leider haben zahlreiche Teilnehmer mit ihren Rohdaten nicht am Baum von Yfull teilgenommen, so dass diese Information nicht überall vorhanden ist.
Hiermit möchte ich nochmal darum bitten, am Baum von YFull.com teil zu nehmen und Eure Daten zu übermitteln. Für mich ist das der schönste Baum und die Arbeit mit den Werkzeugen, welche er bietet, hat mir sehr geholfen das Thema besser zu verstehen. Wenn Ihr selber keinen Nutzen darin sieht, dann könnt Ihr das ja auch einfach nur für mich machen. Ich wäre Euch auf jeden Fall sehr dankbar.
Pingback: Y-Haplogruppen und Migration, am Beispiel von I-L38>BY14026 – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT