Wollte man die Y-DNA zweier Männer vergleichen, griff man bisher zu Y-STR Tests mit möglichst vielen Markern. Hat man einen, oder mehrere NGS (Next Generation Sequencing) zur Verfügung, so bietet sich eine neue Methode, wie man die Verwandtschaft zweier Männer, in rein väterlicher Linie, anhand von „jungen“ SNPs bestimmen kann. Deine Jüngsten SNPs sind jene, welche nur in Deiner Probe festgestellt wurden. Diese werden „Novel SNPs“ oder „private SNPs“ genannt.
Möglich wird diese Methode durch die Firma YSEQ.net und der Möglichkeit jede SNP, welche mit dem Sanger Verfahren Testfähig ist, für wenig Geld auch tatsächlich testen zu können, nachdem man sich diese „wünscht“. Diese Variante funktioniert jedoch nur, wenn die Anzahl der privaten SNPs, bzw. der SNPs im terminalen Block nicht zu hoch ist, da mit zunehmender Anzahl der zu testenden SNPs die Kosten hierfür steigen.
- Nachdem man einen NGS gemacht hat, sollte man einige Vorbereitungen treffen.
- Wurden die Vorbereitungen getroffen, kann ein weiterer Tester schnell und kostengünstig die Verwandtschaft feststellen und die jüngste gemeinsame Y-SNP bestimmen (Neue gemeinsame terminale SNP).
1. Vorbereitungen nach erfolgtem NGS Test.
1.1 SNPs aus NGS in Liste eintragen.
Eine vernünftige Sortierung dieser Y-SNPs nach alter ist nur möglich, wenn es, aufgrund mehrerer Tester in dieser Y-Haplogruppe, mehrere Verzweigungen gibt. Abb. 1 zeigt die SNPs von BY25359>BY25363>BY35090>Y130323 in vier Blöcken. Die Reihenfolge innerhalb der Blöcke kann nicht festgestellt werden, bevor eine weitere Verzweigung hinzukommt, indem ein Tester nur für einen Teil der SNPs positiv ist. Die jüngsten SNPs sind in Block A, die älteste in Block D.

Legt eine Liste mit Euren „jüngsten“ SNPs an. Als Spalten nehmt Ihr: Position hg38, ancestral allele, derived allele, Region, SNP-Name, Y-Haplogruppe, YSEQ und nach Belieben weitere Spalten, mit für Euch wichtige Informationen. In die Spalte „Region“ könnt Ihr folgende Formel eintragen (Spalte A = Position hg38):
=WENN(A2<2781479;”PAR1″;WENN(A2<10072350;”ok”;WENN(A2<11686750;”CEN”;WENN(A2<20054914;”ok”;WENN(A2<20351054;”DYZ19″;WENN(A2<26637971;”ok”;WENN(A2<26673211;”PostPali”;”PAR2″)))))))
Spalte YSEQ:
- YSEQ empfiehlt nur SNPs außerhalb der hg38 Regionen PAR1, CEN, DYZ19, PostPali und PAR2 zu testen, da diese nicht Sinnvoll für phylogenetische Studien sind. SNPs aus den anderen Regionen könnt Ihr als „SNP wünschen“ kennzeichnen.
- SNPs aus den hg38 Regionen PAR1, CEN, DYZ19, PostPali und PAR2 als „nicht bestellbar“ markieren.
- Überprüft mit der Suche von YSEQ.net, welche der SNPs bereits „bestellbar“ sind. Bitte beachtet, dass einige SNPs mehrere Benennungen haben (z.B. BY35090 = Y129739).
1.2 Strategie überlegen und SNPs zur Bestellung „wünschen“
Diese Methode kann in unterschiedlichen Fällen angewandt werden. Es macht Sinn, sich erst mal Gedanken zur individuellen Strategie zu machen und zu überlegen, welche SNPs für Eure Studien bestellfähig sein sollen. In den meisten Fällen dürften dies die privaten Varianten (Block A, Abb.1), sowie Varianten aus dem terminale Block (Block B, Abb. 2) sein.
1.2.1 Wish a SNP
Mit „Wish a SNP“ teilt man YSEQ mit, welche SNPs in das Sortiment aufgenommen werden sollen. Dabei muss man nur berücksichtigen, dass diese in den testfähigen Bereichen liegen.
- „Haplogroup“ und „Reference Sequence“ (hg38/GRCh38) in den Optionen auswählen.
- >>Add to Cart.<<
- Anzahl der SNPs eintragen.
- >>Checkout<<
- Unter „Add Comments About Your Order” folgende
Informationen aus Eurer Liste eintragen:
- Name der SNP (Falls bereits benannt)
- Position hg38
- Ancestral allele
- Derived allele
- Haplogruppe für diese SNPs
- Bestellen
Wenige Tage später solltet Ihr die Information erhalten, welche SNPs bestellbar sind und welche nicht. Tragt diese Informationen in Eure Liste ein.
1.2.2 Wish a Panel
Bei mehr als sieben SNP kann man mit „Wish a Panel“ die SNPs in Pakete bündeln. Dies erleichtert die Handhabung beim Bestellvorgang im Vergleich zum Bestellen der einzelnen SNPs.
- „Haplogroup“ in den Optionen auswählen.
- >>Add to Cart.<<
- Anzahl des Panels eintragen.
- >>Checkout<<
- Unter „Add Comments About Your Order” folgende
Informationen aus Eurer Liste eintragen:
- Name des Panels
- Liste der SNPs
- Kurze Beschreibung zum Panel
- Für wen ist dieses Panel gedacht?
- Bestellen
Wenige Tage später kommt die Bestätigung, dass das Panel bestellbar ist.
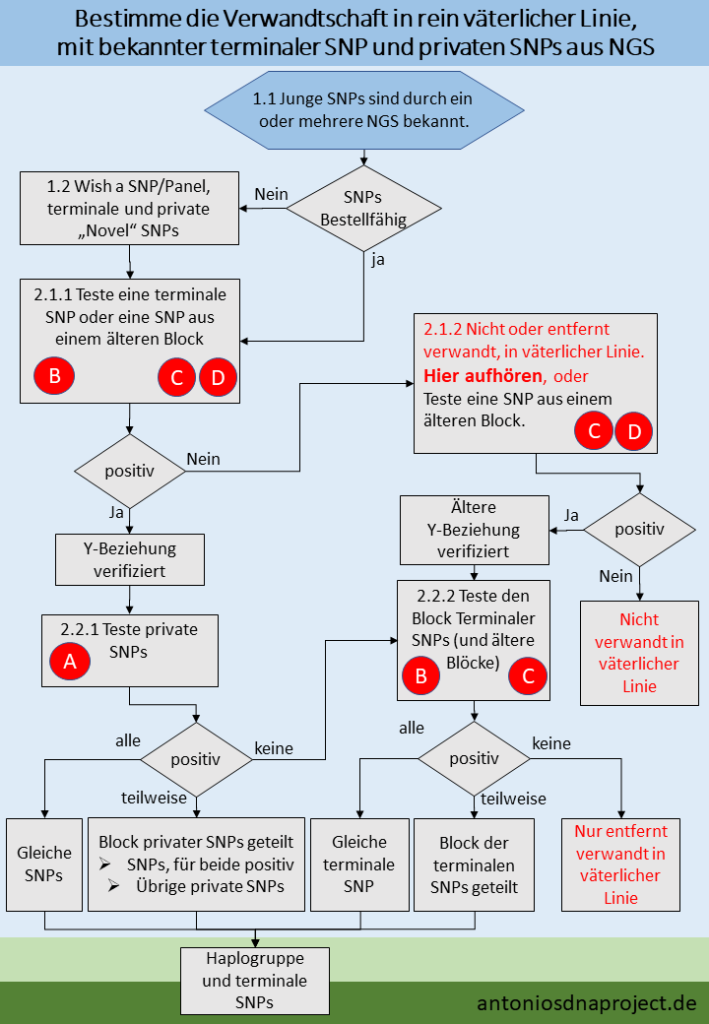
2. SNPs bestellen
2.1 Prüfen ob eine Verwandtschaft besteht
- Man kann nun eine einzelne SNP testen und damit prüfen, ob eine Verwandtschaft in rein väterlicher Linie besteht. Man nimmt hierfür entweder eine terminale SNP oder eine SNP aus einem älteren Block.
- Vorsicht. Nimmt man eine zu junge terminale SNP, kann es sein, dass der MRCA (Most Recent Common Ancestor) älter ist, als die getestete SNP. Ob dies der Fall ist kann man durch testen einer SNP, eines älteren Blocks, überprüfen.
2.2 Nähe der Verwandtschaft bestimmen
- Hat man festgestellt, dass eine Verwandtschaft besteht, testet man die privaten SNPs. Von diesen sollten dann einige positiv und andere negativ sein. Es ist aber auch möglich, dass keine oder alle positiv sind. Anhand der Anzahl der nicht übereinstimmenden SNPs (negative SNPs) kann man den Grad der Verwandtschaft schätzen.
- Sind alle privaten SNPs negativ, besteht die Möglichkeit, dass ein Teil der terminalen SNPS auch negativ sind. Dies sollte man prüfen.
3. Vorteile dieser Methode
- Einfachste und günstigste Lösung zum Verifizieren von Y-Beziehung. (falls „junge“ Terminale SNP aufgrund eines NGS-Tests bekannt und niedrige Anzahl an philoequivalenten und private SNPs.
- Nahezu jede SNP bestellbar „Wish a SNP“.
- Erstelle benutzerdefinierte Panels.
- Teile Blöcke von privaten „Novel“ oder philoequivalenten SNPs (z.B. Block Terminale SNPs) ohne weiteren NGS, zur näheren Bestimmung einer Y-Beziehung.
- Probe wird für Erweiterungen eingelagert.
- Kaufe ein leeres DNA Sample Kit und bestelle das Produkt Deiner Wahl, sobald das Kit im Labor eingetroffen ist.
Pingback: YFull – Erste Schritte – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT