Anfang 2017 machte ich einen DNA-Test. Mein Hauptanliegen war es, eine Familienlegende in meiner väterlichen Linie zu erforschen. Ich wollte wissen, ob es stimmt, dass mein Vorfahr in väterlicher Linie tatsächlich ein Mann namens Gerakaris ist, der aus Mani kam (Meine Eltern kommen aus Nordgriechenland, Mani ist in Südgriechenland). Deshalb lag mein Fokus vom Anfang an auf der Y-DNA. Ich bin anfangs sehr naiv an das Thema heran gegangen und war der Meinung, dass mir ein einziger Y-DNA-Test dabei helfen könnte und bestellte einen sogenannten Y37, zusammen mit einem Test für atDNA und mtDNA.
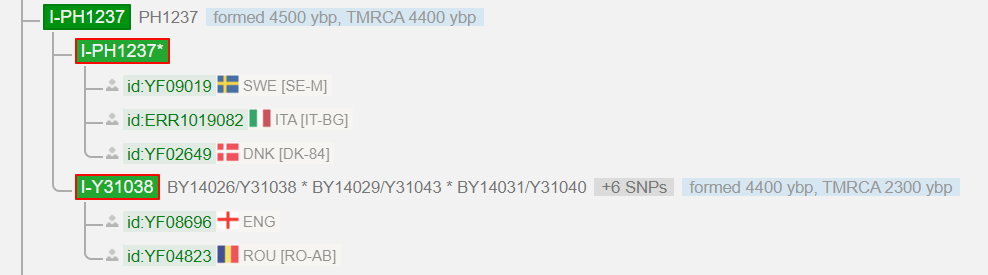
Ich bekam 37 Y-STR Marker, mit denen ich nicht viel anfangen konnte und eine Vorbestimmung für eine Y-Haplogruppe. Diese war I-M170, mit einem TMRCA (Time to Most Recent Common Ancestor) von 27500 ybp. Der Anbieter FTDNA ist dafür bekannt, dass die Vorbestimmungen sehr konservativ sind. Diese Marker wurden im Anschluss von einem Haplogruppen-Administrator als I-L38 > S2606 > PH1237 (TMRCA = 4400 ybp) interpretiert, eine Haplogruppe, die hauptsächlich nördlich der Alpen vorzufinden ist. Außerdem vermutete er, ich könnte der Erste aus Griechenland sein, welcher zu diesem kleinen südosteuropäischen Zweig gehört. Dies wurde alsbald mithilfe eines SNP-Packs bestätigt. Meine bestätigte Haplogruppe war nun I-L38 > S2606 > PH1237 > BY14026/Y31038 (TMRCA = 2300 ybp). War das wirklich alles? Wie kam diese Haplogruppe überhaupt von Nordeuropa nach Griechenland? Der Y-Baum von YFull endete zu diesem Zeitpunkt bei Y31038, und es saßen gerade mal drei Männer auf diesem Ast: Zwei Engländer und ein Mann aus Rumänien. Ich fragte mich: “Gibt es denn keinen Test, welcher weiter geht, so dass man den Bäumen weitere Äste hinzufügen kann?”
Die Antwort war „Ja, den NGS-Tests“.
Dieser Beitrag vertieft das Thema NGS. Eine Übersicht über die Möglichkeiten zum Testen der Y-DNA findet Ihr im Beitrag Y-DNA, Haplogruppe und Genealogie.
1. NGS – Next Generation Sequencing
Es gibt zwei Arten von NGS (Next Generation Sequencing) Tests. Solche wie den Big Y von FTDNA oder den YElite von FGC, welche zielgerichtet nur die genealogisch relevanten Regionen des Y-Chromosoms auslesen (grüner Kasten, Abb. 2) und den WGS, (Whole Genome Sequencing) welcher wie der Name schon sagt, das gesamte Genom ausliest, einschließlich der gesamten mitochondrialen und autosomalen DNA.
1.1 Targeted NGS
Der Big Y von FamilyTreeDNA (FTDNA) erfreut sich trotz seines verhältnismäßig hohen Preises einer großen Beliebtheit, da man vorhandene Tests, wie den Y37 erweitern kann, ohne erneut eine Probe einzusenden. Zusätzlich verfügt FTDNA über eine große Datenbank, einen eigenen Y-Haplobaum und bietet einige kleine Werkzeuge zum Interpretieren der Ergebnisse.
Der YElite von Full Genome Corporation (FGC) beinhaltet zusätzlich die mt-DNA und die Analyse der Daten.

1.2 WGS
WGS werden von unterschiedlichen Firmen angeboten. Diese beinhalten neben der Y-DNA noch die gesamte mitochondriale und autosomale DNA. Firmen wie Full Genome Corporation (FGC) und YSEQ haben sich auf die genealogische Verwendung von WGS-Rohdaten spezialisiert und bereiten diese dementsprechend auf, so dass ich diese Firmen für Leute empfehlen möchte, welche selber nicht in der Lage sind, diese Schritte zu erledigen. Wenn man sich das selber zutraut oder jemanden kennt, welcher einem behilflich ist, kann man aber auch jede andere Firma wählen, welche WGS anbietet, vorausgesetzt man bekommt die Rohdaten in den Formaten FASTQ oder BAM.
Einen Vergleich der unterschiedlichen NGS bezüglich Abdeckung und deren Bedeutung für die Altersbestimmung findet man hier: ydna-warehouse.org/statistics
1.3 Was bekommt man?
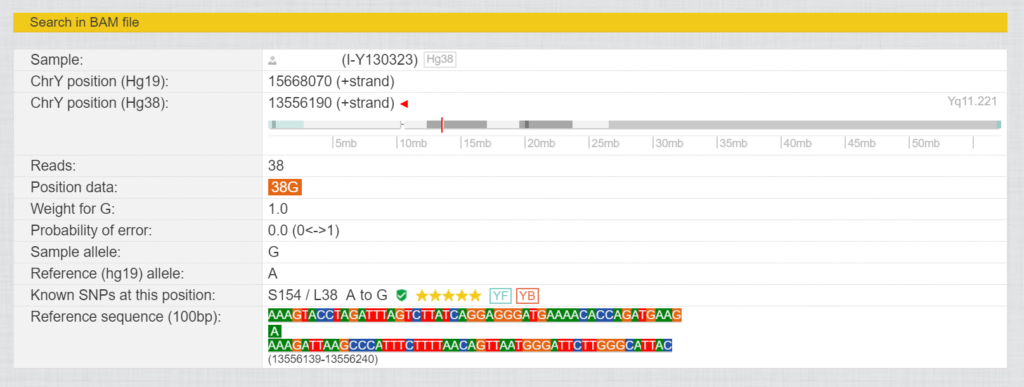
Bei diesen Tests wird nicht nur eine kleine Auswahl des Y-Chromosoms, sondern, je nach Anbieter und Test, der ganze genealogisch relevante Bereich mit über 23 Millionen Basenpaaren getestet. Die erhaltene Sequenz wird an der Human Genome Referenz ausgerichtet, und die Werte des Testers werden mit diesem verglichen. Die aktuelle Version ist HG38, jedoch wird gelegentlich noch die ältere Version HG19 verwendet. Für jedes dieser Basenpaare gibt es eine Nummer für die Position (Diese unterscheiden sich von HG19 zu HG38) und die ermittelten Werte für diese Position in Form von Buchstaben (A, T, G und C). Unterscheidet sich eine solche Position von der Referenz (“Sample allele” ist nicht gleich “Reference allele”), so spricht man von einer SNP (Single Nucleotide Polymorphism). Abb. 3 zeigt die SNP L38, wo es bei der Position 13556190 (HG38) eine Veränderung von A zu G gab.
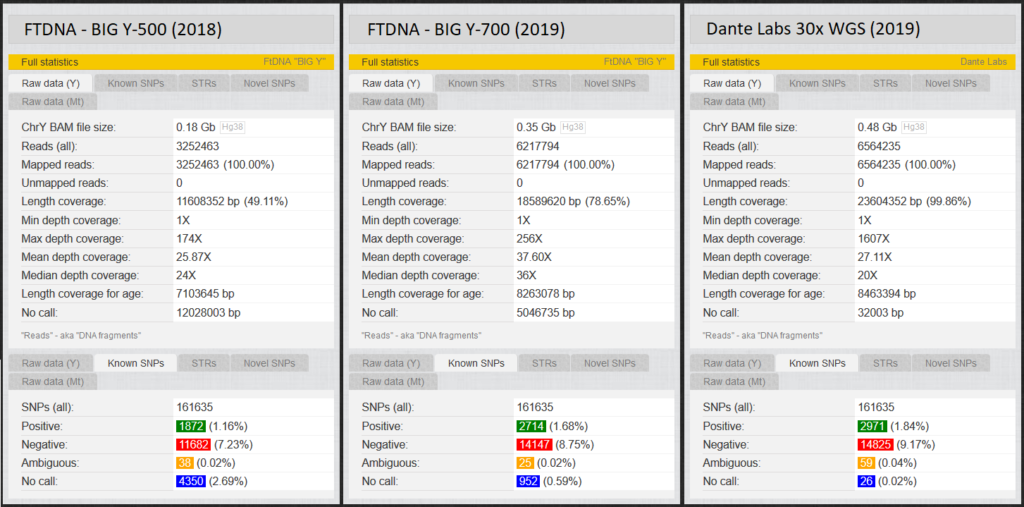
Auf Abb. 4 sieht man unter “Length Coverage“, dass in Abhängigkeit des Tests unterschiedlich viele dieser etwa 23 Millionen Basenpaare getestet werden. „Depth coverage“ gibt hingegen an wie oft die Positionen gelesen wurden. Bei Yfull sind knapp über 160.000 dieser Basenpaare als SNPs bekannt. Jeder von uns Männern dürfte in etwa 2.300 positive Y-SNPs haben. (YFull zählt einige SNPs mehrfach, wenn für diese unterschiedliche Namen existieren. Deshalb 3000 > 2300). Position, welche bei einem Test nicht gelesen wurden, nennt man „no calls“. Hier ist dementsprechend nicht bekannt, ob für diese Variante ein positiver oder ein negativer wert vorliegt, so dass ein Test mit möglichst großer Abdeckung (Length coverage) und wenig no calls angestrebt wird. Wir werden uns später an einem Beispiel die Auswirkungen von diesen no calls ansehen.
Zusätzlich lassen sich aus NGS-Tests mehrere hundert STRs (Short Tandem Repeat) extrahieren und deren Vorteile nutzen. Bei Yfull sind das bis zu 780.
1.4 Rohdaten
1.4.1 FASTQ:
In dieser Datei befinden sich die gesamten Rohdaten, die noch nicht an einer Referenz ausgerichtet sind. Fehlt einem die „richtige“ BAM, so kann man sie mit Hilfe der FASTQ selbst erstellen. Die Programme hierfür laufen unter dem Betriebssystem Linux und das Erstellen dauert mehrere Tage. Alternativ kann man diese Aktion auch bei Sequencing.com oder kostenlos bei usegalaxy.eu durchführen. Hier ist zu beachten, dass dies nur mit schneller Internetverbindung vernünftig ist, da die FASTQ über 60 GB groß sein kann.
1.4.2 BAM:
Dies ist die Datei, welche für die Teilnahme an den Bäumen benötigt wird. In dieser befinden sich die an der Referenz ausgerichteten Daten. Hat man seinen NGS bei einer Firma, wie FTDNA, FGC oder YSEQ gemacht, welche sich auf die genealogische Anwendung des NGS spezialisiert haben, bekommt man einen Link, welchen man einfach nur bei der Bestellung bei YFull angeben muss. Hier kann man den restlichen Absatz ignorieren.
Für alle, welche ihren WGS, bei einer anderen Firma gemacht haben, ist
folgendes von Bedeutung:
Die aktuelle Version der Referenz
ist HG38, jedoch wird gelegentlich noch die ältere Version HG19 verwendet. Die
bevorzugte Version für das Übertragen an YFull ist die HG38, man kann aber
zur Not auch die HG19 verwenden, wenn man
nicht die HG38 zur Verfügung hat, oder nicht in der Lage ist, diese selber aus
der FASTQ zu erstellen. Im selben Verzeichnis wie die BAM, sollte sich
eine Datei mit der Endung BAI befinden. Diese ist die Index Datei.
Eine BAM aus einem WGS enthält, neben der Y-DNA, die komplette mtDNA und atDNA und ist bis zu 100 GB groß. Zur Teilnahme an YFull wird aber nur ein kleiner Teil davon benötigt, die Abschnitte der Y-DNA und der mt-DNA. Hat man Erfahrungen mit Linux, kann man mit dem Programm Samtools eine Datei erzeugen, welche nur diese enthält. Man muss sozusagen die atDNA rausschneiden. Nutzer von Windows können zu einem Werkzeug namens WGS Extract (Link zum Download) greifen. Damit kann man diese Datei intuitiv erstellen und später über die eigene Cloud zur Verfügung stellen. Mit WGS Extract kann man aber auch eine FASTA Datei mit der mtDNA erzeugen, sowie die atDNA extrahieren und in unterschiedlichen Formaten speichern.
Eine Beschreibung des Programms WGS Extract gibt es hier in einem englischen Blog: http://www.beholdgenealogy.com/blog/?p=3018
1.4.3 VCF:
Das Format VCF erhält nur einen Teil der Rohdaten. Hier ist schwierig eindeutig zu sagen, was genau in dieser Datei enthalten ist, da dies vom Hersteller abhängt. Den schönsten Spruch, den ich hierzu gelesen hatte war: „Traue keiner VCF, die Du nicht selber manipuliert hast“.
Nachdem es seit 1. November bei FTDNA kostenpflichtig geworden ist, eine BAM erzeugen zu lassen, hat YFull entschieden, zukünftig auch die vcf-Datei von FTDNA zur Teilnahme am Baum zuzulassen.
Hat man, nachdem man einen WGS machte, die BAM noch nicht zur Verfügung, kann man aus dieser einige Y-Informationen auslesen. Mit dem DNA Kit Studio kann man die Y-Information aus der VCF auslesen und mit dem Morley Predictor analysieren.
1.5 Vorteile von NGS
- Alle positive SNPs werden ermitteln, inclusive der „privaten“ Varianten, welche vorher bei noch keinem anderen Probanden positiv getestet wurden.
- Extrahieren von mehreren hunderten Y-STR.
- Feststellung und Bewertung der väterlichen Verwandtschaft zwischen zwei Männern per Y-SNP, Y-STR und privaten SNPs.
- Aktives Mitwirken beim „wachsen“ der Y-Bäume, wie das Formen neuer Zweige.
- Ermitteln der Y-Haplogruppe und Untergruppe für vor- und frühgeschichtliche Forschungen, aber auch Migrationsbewegungen der letzten Jahrhunderte.
- Hochladen der Rohdaten zu Yfull.com.
- Automatische Aktualisierung der terminalen SNP durch Hinzufügen neuer Probanden zu den Datenbanken (Bäumen).
- „private Varianten“ und junge terminale SNP zum Verifizieren von väterlichen Beziehungen, wie in Punkt 4.0 von Beitrag Y-DNA, Haplogruppe und Genealogie beschrieben.
2. Der Y-Baum
2.1 Die Y-Haplogruppen Bäume:
- Wenn man seinen NGS bei FTDNA gemacht hat, wird man nach Fertigstellung des Tests im hauseigenen Y-DNA Haplobaum plaziert.
- Tester der Y-Haplogruppe R1b können ihre Rohdaten bei The Big Tree von Alex Williamson hochladen.
- Beim Y-Baum von https://yfull.com/ kann jeder Tester, unabhängig von der Y-Haplogruppe und dem Anbieter des Tests, mit seinen Rohdaten teilnehmen. Dieser Baum ist mein Favorit.
- Der Baum von ISOGG wird regelmäßig aktualisiert.
2.2 Eine Linie am Y-Baum von Adam bis Antonios
Wie schaut solch eine Linie auf dem Baum aus? Die 2300 positiven SNPs, welche man bei einem NGS erhält, kann man sich als eine Kette von Veränderungen vorstellen, die zwischen dem sogenannten Referenz-Chromosom („Adam“) und dem Tester entstanden sind. Man kann in etwa davon ausgehen, dass sich alle 80 bis 100 Jahre eine SNP verändert hat. Die Abbildung zeigt die ersten 1.949 SNP, welche von „Y-Adam“ bis hin zu dem MRCA (Most Recent Common Ancestor) von I-M170, in den Blöcken AO-T > A1 > A1b > BT > CT > CF > F > GHIJK > HIJK > IJ > I-M170, entstanden sind. Das sind sehr alte SNPs, welche vor vielen Jahrtausenden entstanden sind. Alle Männer, welche zur Y-Haplogruppe gehören, sind positiv für diese knapp 2000 SNPs.
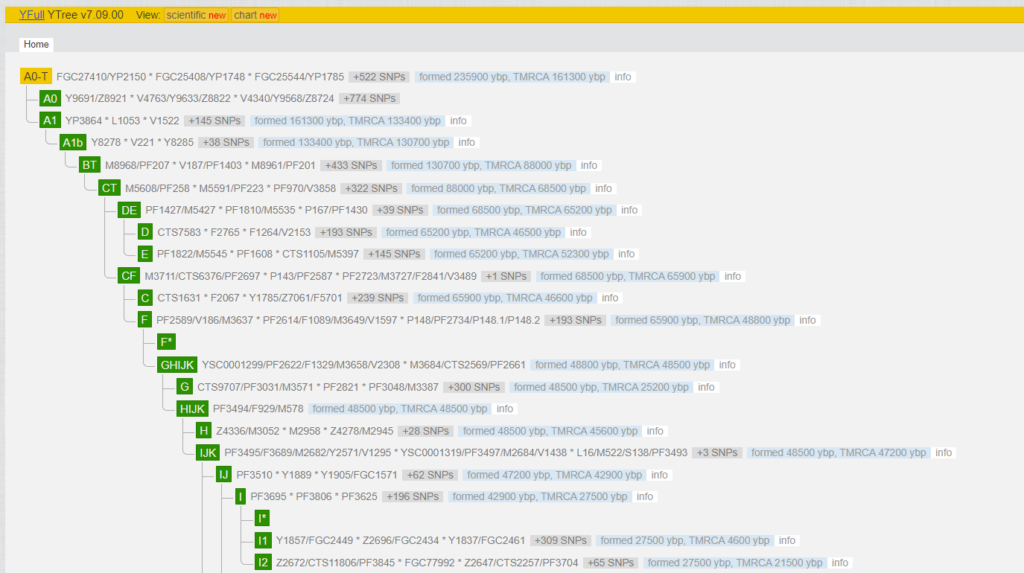
Der Ausschnitt ist für I-M170. Den gesamten Baum findet man unter YFull in unterschiedlichen Ansichten (classic, scientific und chart). Es lohnt sich, diesen etwas genauer anzusehen. Die restlichen 348 SNPs sind zwischen dem MRCA von I-M170 und dem MRCA meiner Untergruppe I-Y130323 entstanden. I2-M438 > CTS2257 > L460 > M436 > Y10705 > L38 > S2606 > Y13067 > Y13076 > PH1237 > Y31038 > BY25359 > Y125026 > Y128714 > Y130323. Hier sieht man die grobe Reihenfolge der SNPs, denn die Reihenfolge innerhalb dieser Blöcke ist (noch) nicht bekannt.

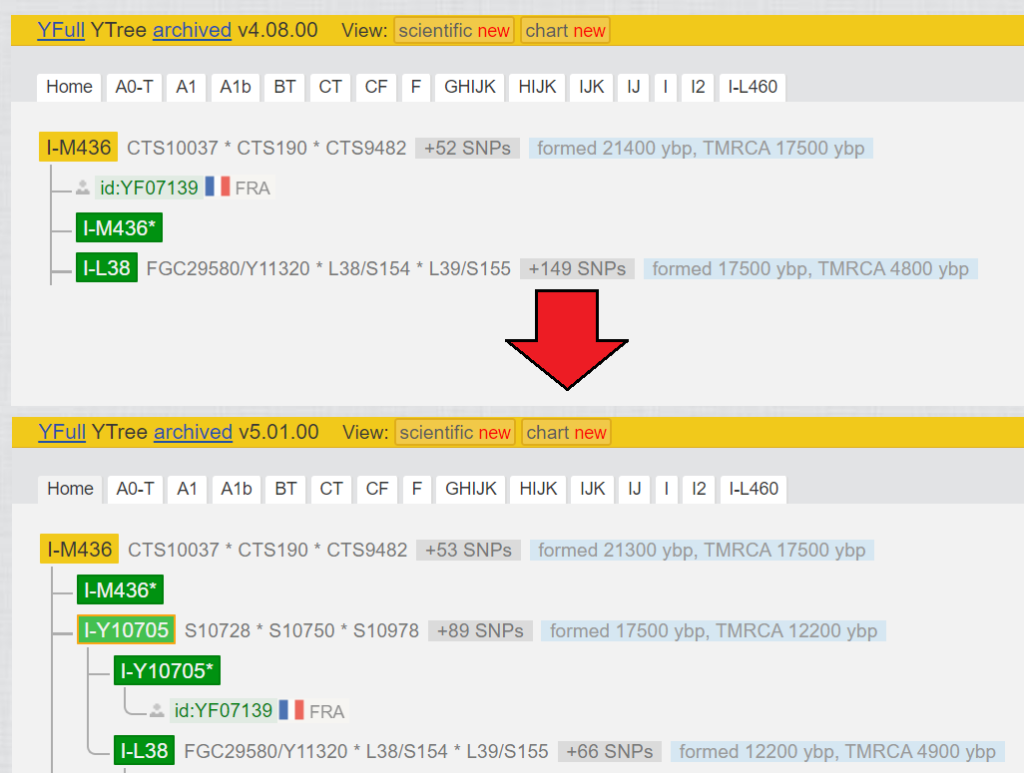
Bis zur SNP Y31038 (BY14026) habe ich alle Kits aus der Ansicht entfernt, damit man ein besseres Bild von dieser Linie bekommt. Das ist genau die SNP, welche ursprünglich durch das SNP-Pack ermittelt wurde. Was man unterhalb sieht, wäre mir ohne NGS verborgen geblieben. (Man beachte den Vergleich zum Stand von April 2017, Abb. 1) Das ist für mich der interessante Teil – Die „jüngsten“ 26 Varianten (=SNPs), welche sich in den letzten zweitausend Jahren verändert haben. Diese sind auf die vier Blöcke BY25359 > Y125026 > Y128714 > Y130323 aufgeteilt und werden für weitere Untersuchungen verwendet. (Die Kits unter Y130323 haben keine nennenswerten „privaten“ Varianten.)
Meine Linie ist nur eine von vielen. Ein Baum wird das Ganze erst, wenn mehrere Linien miteinander verknüpft werden. Ich hoffe, dass in den nächsten Jahren die Preise für das WGS fallen und mehr Männer an Y-Baum-Projekten teilnehmen und die Bäume noch „schöner“ wachsen. Auch wünsche ich mir, dass die Menschen, welche einen WGS aus anderen Gründen machten, mit ihren Rohdaten am Projekt von YFull.com teilnehmen und die Phylogenetischen Forschungen unterstützen.
2.3 Was passiert, wenn man an einem Baum teilnimmt?
Man nimmt an einem Y-Baum teilt, indem man z.B. den NGS von FTDNA durchführt und somit auf dessen Haplobaum kommt, oder die Rohdaten eines NGS (auch WGS), unabhängig vom Hersteller, zu YFull oder „The Big Tree“ übermittelt. In den Datenbanken werden Eure Ergebnisse mit den vorhandenen Teilnehmern verglichen, und Eure Linie, über den Baumstamm bis hin zu Eurem Zweig, wird in den Baum integriert.
Der größte Teil Eurer Varianten sind in der Datenbank bereits bekannte SNPs. Anhand dieser wird Euch ein vorläufiger Platz auf dem Y-Baum zugewiesen. Zusätzlich habt Ihr noch einige „neue“ SNPs, oder auch „private“ SNPs genannt, welche vorher bei noch keinem anderen Tester gefunden wurden. Nach der genaueren Analyse werdet Ihr Eure genaue Position im Baum einnehmen. Es gibt unterschiedliche Situationen, die sich einstellen können. Diese will ich Euch Mithilfe von Abb. 6 erklären. (Punkte 2.3.1 bis 2.3.3)
2.3.1 Teilen aller SNPs:
YF63639 und YF65688 (YF15460 vor Upgrade) sind für alle Varianten positiv, für die auch YF11483 positiv ist. Die Teilnehmer haben, was ihre SNPs betrifft, die gleiche Y-DNA. Keiner der Teilnehmer hat private SNPs übrig. Deswegen wird die TMRCA für diese Gruppe auf 50 „years before present“ geschätzt. (Die tatsächliche TMRCA beträgt ca. 150 bis 200 Jahre.)
Trotzdem die SNPs die gleichen sind, kann es sein, dass es einige Unterschiede bei den STRs gibt. Ist dies der Fall, kann man diese zur weiteren Gruppierung unterhalb des MRCA nutzen.
2.3.2 Bilden einer generischen Untergruppe:
YF19586 Ist positiv für alle SNPs innerhalb des Blocks von Y125026 (Gilt für alle Y125026*). Er teilt mit keinem „private“ Varianten und ist sozusagen der einzige Teilnehmer einer zukünftigen Untergruppe. In solchen Fällen kennzeichnet man die Kandidaten einem Stern „*“.
Ein möglicher Grund dafür, dass keine Varianten für Untergruppen gefunden wurden kann sein, dass diese Aufgrund einer schlechten Abdeckung der Tests (z.B. Big Y-500) und dadurch erhöhten no calls, bei dem einen oder anderen nicht mitgetestet wurden. Diese Situation kann durch das Erweitern auf einen höherwertigeren Test, wie ein WGS oder ein Big Y-700, verbessert werden. Es gibt aber keine Garantie, dass neue private SNPs gefunden werden. Im Fall von 2.3.1 habe ich trotz Erweiterungen keine privaten Varianten ermitteln können.
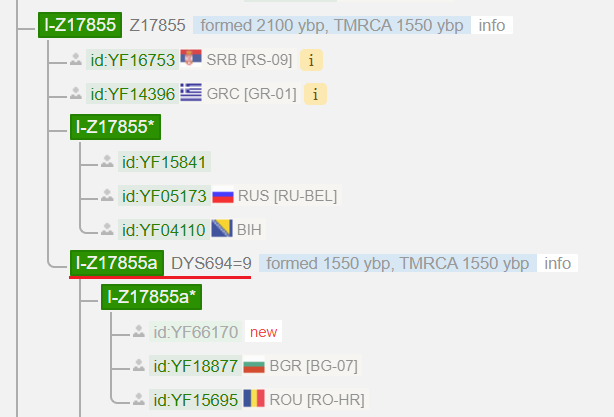
(Ein Blick auf die Y-STRs lässt vermuten, dass man die Teilnehmer von Y125026 aufgrund einer bestimmten STR (DYS448, 4 Sterne) in zwei Gruppen unterteilen könnte. Siehe 2.3.6)
2.3.3 Teilen einiger privater Varianten:
YF65013 hatte mehr Glück. HGDP00894 (Teilnehmer einer wissenschaftlichen Studie) war bereits vorher in der Datenbank, als Y125026*. Für sieben ehemals private SNPs von HGDP00894 ist YF65013 positiv. Diese bestimmen nun die neue Untergruppe Y177573.
2.3.4 Einzelne SNPs aus einem Block lösen:
Als YF63287 hinzukam, gab es die Untergruppe Y125026 noch nicht. Diese war damals noch als BY25359 bekannt. Alle Teilnehmer dieser Untergruppe waren sowohl positiv für die Varianten, welche nun unter Y125026 stehen, als auch für BY25359.
YF63287 ist nur für BY25359 positiv, aber negativ für die SNPs unter Y125026. Dadurch wurde der Block BY25359 zu Y125026, indem die SNP BY25359 rausgelöst wurde. Es war nur Zufall, dass für die Benennung des Blocks die älteste SNP gewählt wurde.

2.3.5 Ein Block zweiteilen:
Eigentlich das Gleiche, wie bei Punkt 2.3.4, aber mit drastischer Auswirkung sieht man auf Abb. 8. Der Block der Y-Haplogruppe zählte 152 Varianten, mit einem Flaschenhals von 12700 Jahren, bis YF07139 hinzukam.
- SNPs für die YF07139 negativ ist, kamen in den Block L38.
- SNPs für die YF07139 positiv ist, kamen in den Block Y10705.
- zusätzlich gab es eine Menge SNPs, welche beim Test von YF07139 nocalls waren (altes Big Y-500). Diese konnten nicht akkurat positioniert werden, und wurden auf die Blöcke L38 und Y10705 gleichmäßig verteilt. Diese können erst richtig positioniert werden, wenn entweder YF07139, oder jemand auf gleicher Position Y10705* einen Test mit besserer Abdeckung (Length coverage) hochlädt.
2.3.6 Durch STR definierte Zweige
Bisher ging es nur um Zweige, welche durch SNPs definiert sind. Diese können auch durch STRs definiert sein, welche unterschiedliche Mutationsraten haben. Mutiert eine STR nur sehr selten (Niedrige Mutationsrate), so kann man diese auch für das Unterscheiden und Definieren von Zweigen verwenden. YFull verwendet STRs mit niedrigen Mutationsraten in ihrem Baum, die mit „fünf Sternen“ gekennzeichnet sind.
2.4 Benennung der neuen Varianten und SNP-Blöcke
2.4.1 Benennung der Varianten:
Kommen neue Varianten zum Baum hinzu, werden diese benannt, von demjenigen, welcher sie „entdeckt“ hat. Hier werden je nach „Entdecker“, unterschiedliche Präfixe verwendet. Die momentan geläufigsten sind:
- A = YSEQ.net
- BY = Big Y-500 von FTDNA
- FGC = Full Genomes Corp. (FGC)
- FT = Big Y-700 von FTDNA
- Y = YFull.com
Eine vollständige Liste der Präfixe gibt es bei ISOGG.org
Es kann vorkommen, dass SNPs von mehreren gleichzeitig „entdeckt“ und benannt werden. Diese werden oft mit einem Schrägstrich zusammengeschrieben (z.B. Y128714/BY35091).
2.4.2 Benennung der SNP-Blöcke
SNP-Blöcke werden nach einer beinhaltenden SNP benannt.
Auch hier gibt es keine übereinstimmende Regel und es kann vorkommen, dass auf den unterschiedlichen Y-Bäumen für den gleichen Block unterschiedliche SNPs verwendet werden. Ein gutes Beispiel ist auch hier Y128714. Dieser Block hat gar drei Bezeichnungen:
- YFull = Y128714
- FTDNA = BY35090
- ISOGG = A577

Innerhalb der Blöcke können wir die Reihenfolge der SNPs nicht feststellen. Die für die Benennung verwendete SNP kann somit die älteste, die jüngste, aber auch eine dazwischen sein. Erst, wenn der Block durch einen weiteren Test geteilt wird, ist das möglich.
2.4.3 ISOGG Longform
Einige von Euch kennen die Schreibform für Y-Haplogruppen, welche mit einem Großbuchstaben beginnt und dann abwechselnd aus Kleinbuchstaben und Zahlen besteht (Abb. 10). Nimmt man diese Schreibform, so hat I-L38 momentan I2a1b2a für I2-M438 > CTS2257 > L460 > M436 > Y10705 > L38.
| I | 2 | a | 1 | b | 2 | a |
| M170 | M438 | CTS2257 | L460 | M436 | Y10705 | L38 |
Verwendet man diese Schreibform, sollte man unbedingt das Jahr mit angeben, da diese regelmäßig angepasst wird. Als ich 2017 begann war I-L38 noch I2a2b und nicht lange zuvor I2b2. Diese Schreibweise ist zwar sehr bildhaft, aber ich nutze sie wegen Verwechslungsgefahr nicht gerne.
2.5 Warum unterscheiden sich die Zweige auf unterschiedlichen Bäumen?
Vergleicht man die Y-Bäume von YFull und FTDNA, stellt man fest, dass Zweige oder Abzweigungen fehlen können. Das liegt daran, dass die Datenbanken diese Informationen nur verarbeiten können, wenn sie diese auch bekommen. Die Gründe sind:
- Der Test wurde bei FTDNA gemacht und nicht bei YFull oder „The Big Tree“ hinzugefügt. Das könnte vermieden werden, wenn alle ihren Big Y bei YFull hochladen. Man kann die Tester bitten ihre Rohdaten an Yfull zu übermitteln. Dies mache ich gelegentlich für die Untergruppen von BY25359.
- Fehlt die Information beim Haplobaum von FTDNA, liegt es daran, dass der Test bei einem anderen Anbieter gemacht wurde. FTDNA bietet keine Option zum Hochladen.
3. Unterstützen der Y-Baum Projekte
Seit meinem ersten Y-DNA Test bin ich fasziniert von den Y-Bäumen. Besonders vom YFull Baum und den Haplogruppen I-L38 und I-Y3120. Deswegen unterstütze ich diese Projekte und habe bereits ein paar NGS beigesteuert. Es gibt aber auch ein paar weitere Möglichkeiten, wie man Haplogruppen-Projekte unterstützen kann.
- Teilnahme mit vorhandenen Big Y – Rohdaten an allen Y-Bäumen:
Ich möchte alle, welche einen Big Y machten, ermutigen die Rohdaten auch auf die anderen Y-Bäume zu übertragen. Dies sind die Bäume The Big Tree von Alex Williamson, für Träger der Haplogruppe R1b, und unabhängig von der Haplogruppe der Baum von YFull.com, wo es zwischenzeitlich auch einen mt-Baum gibt. - Teilnahme mit vorhandenen WGS – Rohdaten an allen Y-Bäumen:
Machte man einen WGS wegen der Gesundheitsinformationen, so wird man sich höchstwahrscheinlich eher nicht für dieses Thema interessieren. Unabhängig davon, ob Ihr dafür Interesse entwickeln konntet oder nicht, möchte ich Euch bitten an den oben erwähnten Bäumen (YFull und falls R1b auch The Big tree) eintragen zu lassen. Die Rohdaten habt ihr bereits. Der Grund ist einfach. Einige an Genealogie interessierte Tester warten seit Jahren sehnsüchtig auf Teilnehmer, welche kleine, aber dennoch wichtige Informationen zu ihren Forschungen beisteuern können. So könnten mit Eurer Hilfe z.B. neue Zweige geformt oder SNP-Blöcke geteilt werden.
In meinem Fall warte ich auf Tester, welche Informationen beisteuern, damit meine letzten 26 Varianten sortiert werden können. Außerdem interessiere ich mich für die Haplogruppenverteilung von Griechenland. Besonders interessiert bin ich an den Y-Haplogruppen unter I-M170 der Griechen. - Spenden an Haplogruppen-Projekte:
Bei Haplogruppen-Projekten engagieren sich ehrenamtliche Haplogruppen-Administratoren, welche sich in ihrer Freizeit für die Projekte einsetzen. In diesen Projekten bei FTDNA besteht die Möglichkeit über Group General Fund Contribution Geld zu spenden. Zahlreiche Projekte können hier unterstützt werden (Link ist für I-L38). Meistens wird dieses Geld für das Erweitern des Tests eines Projektteilnehmer verwendet, dessen STR-Marker auf eine bisher unentdeckte alte Untergruppe deuten.

Pingback: Y-Haplogruppe aus atDNA Rohdaten – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT
Pingback: Y-Απλοομάδα από ακατέργαστα δεδομένα atDNA (αυτοσωματικού DNA) – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT
Pingback: Verify relationship in paternal line, with known terminal SNP and private „novel“ SNPs from NGS – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT
Pingback: Bestimme die Verwandtschaft in rein väterlicher Linie, mit bekannter terminaler SNP und privaten SNPs aus NGS – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT
Pingback: YFull – Erste Schritte – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT
Pingback: Y-Haplogruppen und Migration, am Beispiel von I-L38>BY14026 – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT
Pingback: I-M170 und die babylonische Y-Haplogruppen Namensverwirrung – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT
Pingback: Family Finder Y-DNA Haplogruppen – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT