1. Das versteckte Potential von atDNA Tests, für Y-DNA Projekte.
Autosomale DNA Tests (atDNA) erfreuen sich einer wachsenden Beliebtheit. Diese werden meistens mit den Herkunftsanalysen beworben, und bieten zudem Datenbanken für das sogenannte „Matching“ (Der mMn. größte Vorteil dieser Tests). Auf der Seite The DNA Geek gibt es dafür einige Statistiken. Demnach wurden insgesamt fast 30 Millionen atDNA Tests durchgeführt oder hochgeladen. Einige, sind in mehreren Datenbanken vertreten, so dass man die Anzahl der getesteten Männer, sehr grob überschlagen, auf bis zu 10 Millionen Tester schätzen kann. Darin ist ein großes Potential für Y-DNA Projekte versteckt.
Bei den autosomalen DNA Tests von 23andme und Living DNA werden die Y-Haplogruppe und die mt-Haplogruppe direkt angezeigt. Viele wissen nicht, dass in den Rohdaten aller männlicher atDNA Tester, Informationen (Y-SNPs) für die Y-Haplogruppe vorhanden sind, und mit Werkzeugen wie dem Morley Predictor ausgelesen werden können. (Lediglich bei FTDNA werden diese Y-SNPs aus den Rohdaten entfernt).
Unabhängig davon, bei welchem Anbieter man als Mann den atDNA Test gemacht hat, ist es Sinnvoll sich die Ergebnisse genau anzusehen, denn es stecken weitaus mehr Informationen drin, als manche vermuten. Wenn man eine „alte“ Haplogruppe angezeigt bekommt, obwohl jüngere Zweige mitgetestet werden, kann es bedeuten, dass man auf einem seltenen Ast sitzt.
Kein Interesse an der Y-Haplogruppe?
Es kann sein, dass Du selber kein Interesse hast, an der Y-Haplogruppe. Ich möchte Dich trotzdem darum bitten, Dich kurz mit diesem Thema zu befassen, Deine Y-Haplogruppe zu extrahieren und dies zu kommunizieren. So könntest Du jemandem, der sich hiermit intensiv bei einem Y-DNA Projekt beschäftigt, ein sehr wertvolles Puzzlestück beisteuern.
Als Beispiel, eine meiner offenen Fragen: „Gibt es noch einen Griechen mit der Y-Haplogruppe I-L38?“
Meine Y-Haplogruppe ist I-L38, und mein Vorfahr in rein männlicher Linie war aus Griechenland. I-L38 kommt jedoch aus dem Norden Europas und mir war, bis vor kurzem, kein anderer aus Griechenland bekannt, der mit dieser Y-Haplogruppe getestet wurde. Dass mir keiner bekannt ist, bedeutet jedoch nicht, dass es keinen weiteren Tester gibt. Es ist durchaus möglich, dass „der Gesuchte“ einen atDNA Test gemacht hat, mit seinem Ergebnis aber nichts anzufangen weiß. Das ist auch der Grund, warum ich diesen Blog erstellte. Damit mich dieser finden und kontaktieren kann, so dass wir uns über diese Y-Haplogruppe austauchen können.

Abb.1 ist ein Ausschnitt der Übersicht Y-DNA, Haplogruppe und Genealogie
2. Was ist eine Y-Haplogruppe?
Die Y-DNA wird vom Vater nur an die Söhne übergeben. Eine Y-Haplogruppe ist eine Gruppe von Männern, die alle von einem Mann, in rein väterlicher Linie abstammen. Für die Datierung des jüngsten gemeinsamen Vorfahren, der als MRCA (Most Recent Common Ancestor) bezeichnet wird, verwendet man den TMRCA (Time to the Most Recent Common Ancestor) in „years before present“. Bei einer einfachen Bestimmung der Y-Haplogruppe kann der TMRCA bei ungefähr 5000 Jahren liegen. Will man die Haplogruppe „genauer“ wissen, kann man mit zusätzlichen Tests auf Untergruppen verfeinern und kommt unter Umständen auf einen MRCA, welcher vor etwa 1000 Jahre lebte.
Früher verwendete man oft die sogenannte „Lange Form“ für die Benennung von Y-Haplogruppen, welche mit Großbuchstaben beginnt und dann abwechselnd aus Zahlen und Kleinbuchstaben besteht. Mittlerweile nimmt man dafür überwiegend eine SNP (Single Nucleotide Polymorphism) für die alle Gruppenteilnehmer positiv getestet wurden. Meine eigene Y-Haplogruppe ist bekannt als I2a1b2a (Lange Form, gültig für 2019) oder I-L38 (SNP). Jedes Zeichen der Langen form steht für eine SNP.
| Lange Form | I | 2 | a | 1 | b | 2 | a | 2 |
| SNP | M170 | M438 | CTS2257 | L460 | M436 | Y10705 | L38 | S2606 |
| TMRCA (YFull) | 27500 | 21500 | n/a | 20900 | 17100 | 11800 | 4400 | 4100 |
3. Y-Haplogruppe aus den Rohdaten eines atDNA-Tests extrahieren.
Die Y-Haplogruppe wird anhand von SNPs aus der Y-DNA ermittelt. Eine höhere Anzahl getesteter SNPs, kann theoretisch zu einer genaueren Feststellung der Y-Haplogruppe führen. Praktisch ist es jedoch von der Haplogruppe abhängig. (Im Fall von I-L38 bekommt man bei Living DNA, trotz der fast zehnfachen SNPs, gegenüber dem Test von 23andme v5, keine zusätzliche Untergruppe). Man sollte auch wissen, dass es innerhalb der Firmen, aufgrund von „Chipwechsel“ unterschiedliche Versionen gibt, auch bezogen auf die Anzahl von Y-SNPs. My Heritage hat den Chip für dessen Test im Frühjahr 2019 umgestellt. Die Ergebnisse des Tests Version 1 (Bis 03.2019) sind mit Vorsicht zu genießen, da hier nur 482 SNPs getestet wurden, die Version 2 ist aber vergleichbar mit dem Test von 23andme v5.
| Test | FTDNA | My Heritage v1 (Bis ca. 03.2019) | My Heritage v2 (ab ca. 04.2019) | Ancestry | 23andme v3 | 23andme v5 | Living DNA v1 | Living DNA v2 |
| Y-SNPs | 0 | 482 | 3524 | 1729 | 1766 | 3733 | 22500 | 34216 |
Für die folgenden Punkte nehme ich als Beispiel die Rohdaten meiner eigenen Tests, von Ancestry und 23andme v5.
3.1 Rohdaten herunterladen und auf Festplatte speichern.
3.2 Preprocessing, stage 1 of 2
- Auf der Seite des Morley Predictors den Haken setzen, vor „I consent to the processing and collection of my data, as described in the privacy policy“.
- Datei auswählen.
- Inhalt des Kastens “Extracted Y-DNA data“ in eine Tabellen- oder Textdatei kopieren.
- Vergewissere Dich, dass in dem Kasten „Errors encountered during processing“, „no errors to report” steht.
- Weiter mit >>Submit<<.
3.3 Preprocessing stage 2 of 2
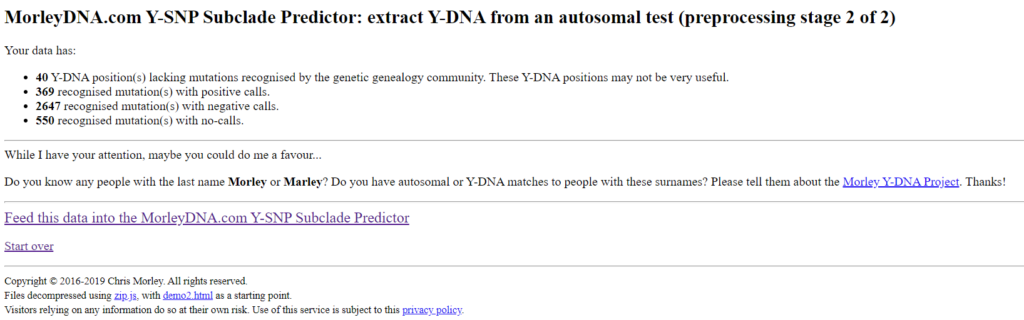
Diese Seite enthält eine Auflistung über die verarbeiteten SNPs und kann als Qualitätskontrolle dienen. Werden hier mehr No-calls (Positionen, die keine Werte enthalten) als positive und negative SNPs angezeigt, muss ein Fehler vorliegen. Bei Tests von My Heritage Version 2, die nach April 2019 durchgeführt wurden, wird eine erhöhte Anzahl an „unrecognised position(s). Are you using data from a source other than AncestryDNA, 23andMe or MyHeritage?” angezeigt. Dies liegt daran, dass der Morley Predictor noch nicht für diesen Test optimiert wurde. Das Ergebnis ist dennoch brauchbar. Die folgenden zwei Abbildungen zeigen die Werte für meine Tests von Ancestry und 23andme v5.
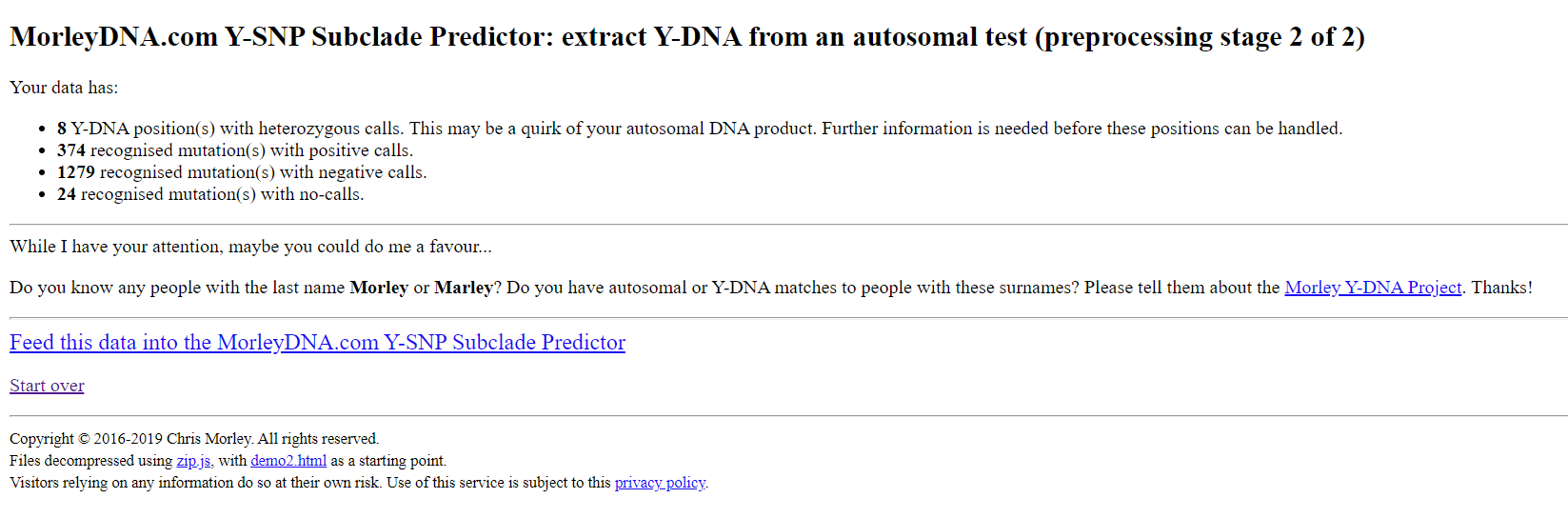
Man erkennt, dass sich die Tests in der Anzahl der positiven, negativen oder No-calls unterscheiden. Es ist interessant, dass der Test von 23andme v5 weniger positive SNPs gefunden hat, obwohl hier doppelt so viel SNPs, im vergleich zu Ancestry, getestet werden. Dafür werden bei 23andme v5 mehr SNPs getestet, für die ich negativ bin, was auch ein Vorteil sein kann.
- Weiter mit >>Feed this data into the MorleyDNA.com Y-SNP Subclade Predictor“<<.
3.4 List of SNPs
- Liste der getesteten SNPs (Inhalt des Kastens „Enter a list of SNP calls, ….“) in eine Tabellen- oder Textdatei kopieren.
- Die Voreinstellung belassen.
- Haken setzen, vor „I consent to the processing and collection of my data, as described in the privacy policy“.
- Haken setzen vor dem reCaptcha, es sei denn Du bist ein Roboter. 😉
- Weiter mit >>Predict<<.
3.5 Das Ergebnis des Morley Predictors
Auf der linken Seite sieht man Vorschläge unter „Suggested terminal clade“. Meistens ist der erste, der mit „most likely“ markiert ist, der richtige, man sollte sich aber alle der Reihe nach ansehen. Wirklich interessant ist jedoch die rechte Seite, wenn man ganz nach unten fährt. (Wichtig! Rechts unten stehen die wichtigen Informationen!) Dort sieht man in einem Bild, anhand der bunten Felder, welche „jüngeren“ SNPs positiv und welche negativ getestet wurden. Abb. 5 zeigt den Test von Ancestry.

- Hinter der Benennung (Lange Form) der Y-Haplogruppen in fetten Buchstaben stehen die SNPs, die zum jeweiligen phylogenetischen Block gehören.
- Die positiven SNPs sind grün, die negativen rot, die No-calls und nicht getestete SNPs ohne Kasten.
- Meine ermittelte Haplogruppe ist I2a2b. Unter dieser Langform war diese Haplogruppe (heute I2a1b2a) im Jahr 2013 bekannt, als Morley diesen Subclade Predictor erstellte (Siehe: ISOGG Y-DNA Haplogroup Tree 2013). Wenn man die Lange Form verwendet, sollte man das Jahr, in dem diese galt, dazu schreiben. I2a1b2a (2019) vom ISOGG Y-DNA Haplogroup Tree 2019, ist das gleiche wie I2a2b (2013). Die Kurzform für diese Haplogruppe ist I-L38.
- Liste oberhalb der Grafik „The suggested classification does not account for the following positive SNPs: “ auf positive getestete, bekannte Untergruppen prüfen. Hier stehen alle SNPs, welche positiv getestet, und in der Grafik nicht berücksichtigt wurden.
- Alternativ, die Liste der getesteten SNPs, welche bei Punkt 3.4 gespeichert wurde, mithilfe eines Y-Baumes z.B. YFull.com untersuchen, ob noch Informationen zu nicht angezeigten Untergruppen enthalten sind.
Als Y-Haplogruppe wurde I2a2b (I-L38) festgestellt (Für diesen Beitrag wird die Lange Form von 2013 verwendet). Folgendes ist in diesem Zusammenhang zu beachten:
- Alle getesteten SNPs hinter I2a2b müssen positiv (grün) sein.
- Alle getesteten SNPs hinter I2a2, I2a, I2 usw. müssen positiv (grün) sein. Diese sind sozusagen die Ahnen von I2a2b und I2a2a (I-M223).
- Alle getesteten SNPs hinter I2a2a müssen negativ (rot) sein. Das ist der „Bruder“, von I2a2b und zweites phylogenetisches Kind von I2a2.
- SNPs, welche diese Regeln brechen, muss man sich gesondert ansehen.
Bei U250/P222/PF3861/S118 handelt es sich z.B. um eine SNP, welche nicht mehr verwendet wird (Siehe https://yfull.com/search-snp-in-tree/), da sie im Baum nicht akkurat platziert werden kann. Die anderen zwei roten SNPs L1005 und CTS3654 wurden bei meinem NGS-Test (Next Generation Sequencing) positiv getestet und sind hier nicht richtig dargestellt.
Es muss sich nicht immer um Fehler handeln. Sind im Block der festgestellten Haplogruppe (bei mir I2a2b) grüne und rote Felder gemischt, so kann das in seltenen Fällen bedeuteten, dass man zu einem sehr seltenen (noch nicht entdeckten) Nebenzweig (z.B. von I2a2b) gehört. Mehr unter 4.2.
Die oben genannten Punkte gelten auch für den Test von 23andme v5, siehe Abb. 6. Vergleicht man die zwei Abbildungen, sieht man, dass teilweise andere SNPs getestet wurden. Ganz unten sieht man, dass die Untergruppe L533 negativ getestet wurde.

Dies ist nicht die einzige Untergruppe, welche von 23andme v5 getestet wird. Meine Y-Haplogruppe, die mir auf der Internetseite von 23andme angezeigt wird, ist I-L38>S2606. Diese SNP (S2606) wird in der Grafik vom Morley Predictor vom Jahr 2013, nicht angezeigt, da diese damals noch nicht bekannt war. Die SNP S2606 erscheint in der Liste oberhalb der Grafik „The suggested classification does not account for the following positive SNPs: “ und ist auch in der Liste von 3.4 als positive SNP zu finden.
In verschiedenen Foren liest man immer wieder die Frage: „Wie tief geht dieser Y-DNA Test?“. In meinem Beispiel geht der Test von 23andme v5 bis I-L38>S2606, mit einem TMRCA von 4100 ybp. (years before present). Abschätzung des Alters von Yfull (yfull.com/tree/I-L38/).
4. Zusätzliche Informationen durch „negative Untergruppen“.
Mit den atDNA Tests werden eine Vielzahl an Y-SNPs getestet. Einige davon werden positiv getestet, andere negativ. Mit den positiven, findet man den Weg zu seiner Y-Haplogruppen-SNP. Dabei kann man übersehen, wie wichtig es ist, die SNPs zu kennen, welche bei einem Test negativ getestet wurden. Besonders dann, wenn der TMRCA für die ermittelte Y-Haplogruppe sehr hoch ist, kann es sehr interessante Gründe dafür geben. Du könntest zu einer alten, seltenen Y-Haplogruppe gehören.
Für die folgenden Punkte wird nur noch der Test von 23andme v5 betrachtet. Man kann die Dateien, welche unter Punkt 3.4 und Punkt 3.2 abgespeichert wurde, zur weiteren Analyse verwenden, um in der Grafik nicht dargestellte SNPs abzufragen.
- Die Liste von 3.4 ist leichter in der Handhabung. Sie enthält alle getesteten SNPs mit einem Plus für positiv und einem Minus für negativ. Wurden keine SNP für die getestete hg19 Position erkannt, wird die hg19 Position angegeben.
- Die Liste von 3.2 enthält die Position hg19 und den Wert des Tests für diese Position. (derived, ist der veränderte Wert, wenn Positiv). Auf der Seite genetichomeland.com kann man für die SNPs die passenden Position hg19 nachsehen.
Beispiel A in der Tabelle steht für “Antonios” und zeigt meine Werte, wie sie unter Punkt 3 ermittelt wurden. Die Beispiele F1 bis F4 sind fiktiv. Wir stellen uns vor, diese SNPs würden bei einem Test so getestet, und interpretieren die Ergebnisse.
| Lange Form und SNPs | Pos. Hg19 | Ref. | derived | A | F1 | F2 | F3 | F4 |
| I2a2 (I-M436) | ||||||||
| L35/PF3862/S150 | 22725379 | C | A | + | + | + | + | + |
| M436/P214/PF3855/S33 | 18747493 | G | C | + | + | + | + | + |
| PF3854/P217/S23 | 7628484 | C | T | + | + | + | + | + |
| P218/S32 | 17493630 | T | G | + | + | + | + | + |
| I2a2a (I-M223) | ||||||||
| L34/PF3857/S151 | 7716262 | A | C | – | – | – | – | – |
| P220/S119 | 24475669 | G | T | – | – | – | – | – |
| PF3858/P221/S120 | 8353707 | C | A | – | – | – | – | – |
| I2a2b (I-L38) | ||||||||
| L38/S154 | 15668070 | A | G | + | + | + | – | + |
| L39/S155 | 16199051 | T | C | + | + | – | – | + |
| L40/S156 | 16202267 | T | C | + | + | – | – | + |
| L65/S159.2 | 16626617 | A | G | + | + | + | – | + |
| I2a2b1 (I-L533) | ||||||||
| L533 | 2887198 | G | C | – | – | – | – | + |
| I2a2b2 (I-S2606) | ||||||||
| S2606 | 22527402 | C | A | + | – | – | – | – |
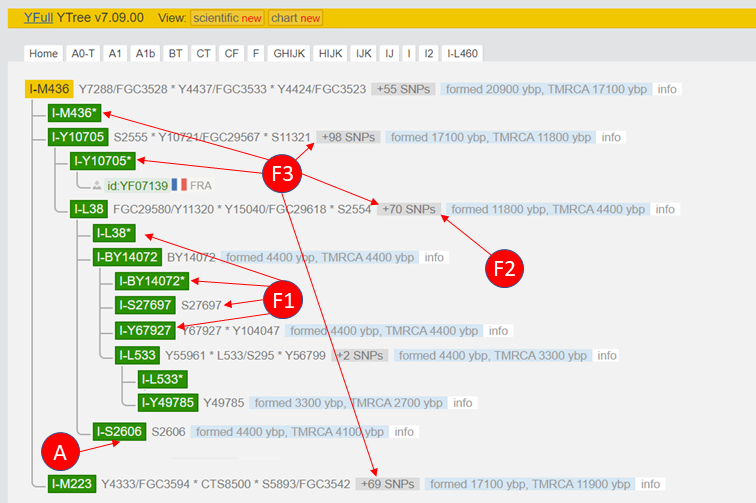
Diese Tabelle beinhaltet nur die SNPs, die von 23andme v5 getestet wurden. Hier könnte man auch die Ergebnisse beider Tests kombinieren. In der zweiten Spalte sind die hg19 Positionen, in der vierten Spalte der Wert für diese Position, so wie diese in den Rohdaten abgespeichert sind. Abb. 7 zeigt die möglichen Positionen am Y-Baum von YFull, in Abhängigkeit von den (fiktiven) ermittelten Werten.
4.1 Beispiel F1: Untergruppen von I-L38 sind negativ.
Beim Test von 23andme v5 werden zwei SNPs für die Untergruppen von I-L38 getestet. Ist entweder S2606 oder L533 positiv, ist das Ergebnis Eindeutig (siehe Beispiele A und F4). Man beachte, dass L533 nicht mehr direkt unter I-L38, sondern eine Untergruppe, der Untergruppe BY14072, ist.
Sind beide SNPs negativ, gibt es drei Möglichkeiten:
- Der Tester ist I-L38*, eine noch nicht entdeckte Untergruppe von I-L38. (unwahrscheinlich, aber Möglich)
- Der Tester ist I-BY14072*, eine noch nicht entdeckte Untergruppe von I-BY14072.
- Der Tester gehört zu einer der Untergruppen von I-BY14072, S27697 oder Y67927.
(Beim Test von Ancestry werden keine SNPs für Untergruppen von I-L38 getestet.)
4.2 Beispiel F2: Ein Teil der SNPs von I-L38 (I2a2b) ist negativ.
Dieser Fall ist auch sehr unwahrscheinlich, aber nicht unmöglich und wäre ein wahrer Glücksgriff für unser I-L38 Haplogruppen Projekt.
Die Y-Haplogruppe I-L38 wird durch 73 SNPs definiert. Vier davon werden beim Test von 23andme v5 getestet. Um zur Haplogruppe I-L38 zu gehören, muss man für alle SNPs dieses phylogenetischen Blocks positiv sein. Ist man negativ für eine oder mehrere dieser SNPs bedeutet dies, dass man den phylogenetischen Block von I-L38 in zwei Blöcke aufteilt. Dies geschah schon mal, als YF07139 (Abb. 7) zum Baum hinzukam. Vorher gehörten alle SNPs der Blöcke I-Y10705 und I-L38 zum Block I-L38. Siehe Blogbeitrag NGS – Nimm Teil am Y-Baum.
Tritt solch ein Fall ein, solltest Du diese Ergebnisse unbedingt jemandem zeigen, der sie genauer bewerten kann. Hierzu kannst Du bei FTDNA die Y-Haplogruppen Projekte durchsuchen und einen Admin der Gruppe kontaktieren, oder bei Facebook einer Gruppe für Deine Y-Haplogruppe beitreten. Im Fall von I-L38 wären dies das I-L38 Haplogruppen Projekt und die Facebook Gruppe I-L38. (Selbstverständlich wäre auch ein Kontakt über diesen Blog möglich).
4.3 Beispiel F3: Alle SNPs von I-L38 (I2a2b) und I-M223 (I2a2a) sind negativ.
Dieser Fall ist, ähnlich dem Fall des Beispiels 3, sehr unwahrscheinlich und deswegen sehr interessant für die Y-Haplogruppen Projekte I-L38 oder I-M223. Die Möglichkeiten in diesem Fall wären:
- Aufteilen des Blocks I-L38, im frühen Stadium
- Aufteilen des Blocks I-Y10705
- Bilden einer Untergruppe mit YF07139.
- Neue unentdeckte Untergruppe unter I-M436
- Aufteilen des Blocks I-M223, im frühen Stadium
Tritt solch ein Fall ein, solltest Du diese Ergebnisse unbedingt jemandem zeigen, der sie genauer bewerten kann. Hierzu kannst Du bei FTDNA die Y-Haplogruppen Projekte durchsuchen und einen Admin der Gruppe kontaktieren, oder bei Facebook einer Gruppe für Deine Y-Haplogruppe beitreten. Im Fall von I-L38 wären dies das I-L38 Haplogruppen Projekt und die Facebook Gruppe I-L38. (Selbstverständlich wäre auch ein Kontakt über diesen Blog möglich).
5. Möglichkeiten zum Erweitern und Vertiefen der Ergebnisse
Die Y-Haplogruppen Informationen, welche man bei einem atDNA bekommt, dürften für viele ausreichend sein. Will man sich mit dem Thema intensiver befassen, kann man die Ergebnisse als Basis nehmen und diese mit unterschiedlichen Produkten erweitern.
5.1 YSEQ Haplogruppen Panel und einzelne SNPs
Eine sehr flexible Produktpalette bietet hierfür die Firma YSEQ aus Berlin. Hier findet man sogenannte „Haplogroup Panels“, aber auch einzelne SNPs, welche zum Testen bestellt werden können (siehe Abb. 1, rechts).
Haplogruppen Panels sind SNP-Pakete mit einer ausgeklügelten Auswahl von SNPs, einer Haplogruppe. Die einzelnen SNPs werden sequenziell getestet, bis man seine terminale SNP erreicht hat. Das ist der Fall, wenn alle bekannten Untergruppen negativ getestet werden. Das für I-L38 passende, wäre das I2 Superclade Panel (Ausschnitt Abb. 8).

Die Panels machen nur Sinn, wenn es genügend Untergruppen gibt, die noch nicht getestet wurden. Das I2 Superclade Panel kostet $99. Eine einzelne SNP kostet $18. Bei einem Superclade Panel rechnet sich das Panel erst ab 6 SNPs. (Haplogruppen Panels für $88 rechnen sich ab 5 SNPs). Hierzu nehmen wir die Beispiele aus Punkt 4.
5.1.1 Beispiel A > Haplogruppen Panel
Beim Beispiel A macht dieses Panel Sinn. Es gibt eine große Anzahl zu testende SNPs. Dieses Panel würde für mich Y31038 als terminal SNP ermitteln. Es gibt aber weitere Untergruppen von Y31038, welche bei diesem Panel nicht getestet werden. Diese Untergruppen kann man im Anschluss testen (Falls vorhanden).
5.1.2 Beispiel F4 > Einzelne SNPs
Beim Beispiel F4 macht das Panel keinen Sinn. Hier würde man gar keine zusätzliche Information bekommen. Man weiß bereits, dass man zu L533 gehört. Die bekannten Untergruppen von L533 sind L533>Y49785 und L533>Y49785>S18432 (siehe Abb. 7 und Y-DNA Haplobaum von FTDNA). Diese zwei SNPs sind bei YSEQ noch nicht bestellbar. Mit dem Produkt „Wish a SNP“ kann man sich wünschen, dass diese SNP in den YSEQ Katalog aufgenommen wird.
5.1.3 Beispiel F1 > Einzelne SNPs
Beim Beispiel F1 macht das Panel keinen Sinn. Die SNPs L533 und S2606 wurden negativ getestet. Es gibt maximal drei SNPs, die zu testen sind. Man geht wie folgt vor:
- SNP BY14072 testen.
- Wenn BY14072 negativ ist, dann ist man L38* (Nur eine SNP wird getestet).
- Wenn BY14072 positiv ist, dann allen Untergruppen, außer L533 (= negativ) testen (S27697 und Y67927).
- Wenn S27697 und Y67927 beide negativ, dann ist man BY14072*.
- Wenn eine der beiden positiv ist, dann ist dies Deine terminale phylogenetische Position.
Ist Dein Ergebnis I-L38*, solltest Du diese Ergebnisse unbedingt jemandem zeigen, der dies genauer bewerten kann. Hierzu kannst Du bei FTDNA die Y-Haplogruppen Projekte durchsuchen und einen Admin der Gruppe kontaktieren, oder bei Facebook einer Gruppe für Deine Y-Haplogruppe beitreten. Im Fall von I-L38 wären dies das I-L38 Haplogruppen Projekt und die Facebook Gruppe I-L38. (Selbstverständlich wäre auch ein Kontakt über diesen Blog möglich).
5.2 NGS (Next Generation Sequencing)
Bei den Beispielen F2 und F3 kann man nicht mit einfachen Mitteln erweitern. Die einzig vernünftige Möglichkeit ist hier ein NGS zu testen und mitzuhelfen, neue Zweige im Y-Haplobaum zu entdecken. Wie dies funktioniert steht im Blogbeitrag NGS – Nimm Teil am Y-Baum.
6. Vorteile, Y-DNA aus atDNA Rohdaten
- Ermitteln der Y-Haplogruppe für vor- und frühgeschichtliche Forschungen.
- Mit diesen Tests kann man zwar nicht feststellen, dass zwei Männer in väterlichen Linie verwandt sind, man kann jedoch feststellen, dass zwei Männer *nicht* in väterlichen Linie verwandt sind, was auch oft hilfreich sein kann.
- Aufgrund der großen Datenbanken und der damit verbundenen autosomalen Treffer (Matche) kann man dies auch gut zum „fischen“ nutzen, sprich Tester finden, welche für die eigenen Y-Forschungen von Belang sind und mit ihnen Kontakt aufnehmen.
Ich habe das mehrfach mit dem cladefinder.yseq.net versucht. Leider bleibt die Website beim Upload meiner Ancestry-Datei immer hängen. Moreley ist mir nicht ganz geheuer da die Daten bis zu 6 Jahre gespeichert werden 🙁
Pingback: Family Finder Y-DNA Haplogruppen – ΑΝΤΩΝΙΟΣ ΔΝΑ PROJECT